科研进展
广州生物院揭示染色质高级结构变化调控细胞凋亡的新机制
真核生物DNA通过缠绕组蛋白八聚体形成以核小体为重复单元的串珠结构,再通过形成远距离的染色质环等高级结构而存储于细胞核中。近年来研究表明染色质高级结构在维持基因表达和细胞命运决定等方面发挥重要作用,且染色质高级结构的形成和维持需要特定转录因子的介导。多功能转录因子CCCTC结合因子(简称:CTCF)是拥有11个高度保守的锌指结构蛋白,除了具有调节基因的转录激活与抑制、基因印记、染色质绝缘、X染色体失活等功能外,还在染色质高级结构调控基因表达方面发挥重要作用,因此CTCF被称为基因组的主编织者。然而CTCF是否存在新的可变剪接体以及该剪切体是否在调控染色质动态变化和细胞命运决定方面有不同于经典CTCF的分子细胞功能仍不清楚。
2019年4月4日,中国科学院广州生物医药与健康研究院姚红杰研究员课题组在国际学术期刊Nature Communications在线发表了题为“An alternative CTCF isoform antagonizes canonical CTCF occupancy and changes chromatin architecture to promote apoptosis”的研究成果。该研究团队发现CTCF存在一个短的可变剪接体(简称CTCF-s);揭示CTCF-s竞争性结合经典CTCF在基因组中的部分DNA结合位点而导致经典CTCF介导的染色质高级结构发生改变,进而改变基因表达和导致细胞凋亡。
运用一系列生物化学和分子生物学的研究方法,该研究团队揭示CTCF存在跳跃外显子3和4的可变剪接体CTCF-s(蛋白序列缺失CTCF的N末端和2.5个锌指结构)。运用带有生物素标签的CTCF和CTCF-s来特异性区分CTCF长和短可变剪接体在基因组DNA结合基序的异同,发现CTCF长和短可变剪接体在基因组上的结合基序虽有很大的相似性,但由于CTCF-s蛋白质缺少锌指结构3(ZF3)(由24个氨基酸组成)的前7个氨基酸(HKCPDCD),因此不再拥有ZF3结合的核心基序(C/G和T/C)。与经典CTCF的DNA结合基序相比,CTCF-s的DNA结合基序有2个碱基的缺失,正好对应CTCF-s中缺失的锌指结构。通过使用特异性识别经典CTCF的N末端的抗体富集CTCF结合的DNA,发现CTCF-s能够竞争性结合经典CTCF在基因组的部分结合位点,导致CTCF/Cohesin在这些结合位点上富集水平明显下降。从染色质高级结构的调控方面,该研究团队发现过表达CTCF-s导致经典CTCF在全基因组范围介导的远距离染色质相互作用能力显著降低。另外该研究揭示CTCF-s可以促进干扰素信号通路相关基因的表达;进一步发现CTCF-s通过破坏经典CTCF在干扰素α诱导蛋白6(IFI6)基因区域的三维空间构象而促进IFI6基因远端增强子和启动子之间的相互作用而激活IFI6基因的表达,从而导致细胞凋亡。
该研究团队阐明了CTCF短的可变剪接体在调节染色质高级结构动态变化方面具有与经典CTCF不同的特征,揭示了CTCF短的可变剪接体在调控细胞命运决定中的新功能。据悉,姚红杰课题组的博士研究生李娇、助理研究员黄开猛博士、博士研究生胡功成是该论文共同第一作者,姚红杰研究员为该论文通讯作者。这项研究得到了来自中国科学院北京基因组研究所杨运桂研究员和南方科技大学Andrew P. Hutchins博士的帮助。该研究得到了来自中国科学院战略性先导科技专项、国家重大科学研究计划以及国家重点研发计划等项目的资助。
文章链接:https://doi.org/10.1038/s41467-019-08949-w
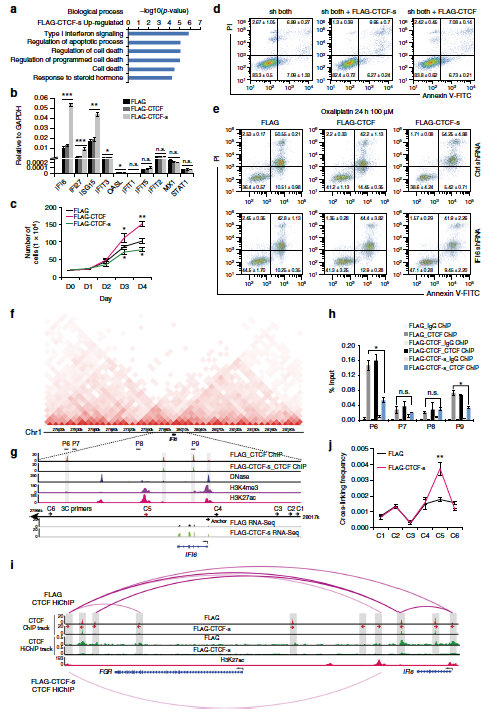
CTCF短的可变剪接体调节经典CTCF在基因组上的结合,导致干扰素相关基因异常激活,从而促进细胞凋亡。
